pcDNA3.1-NgAgo,pET-NgAgo表达质粒载体-替代Cas9的新型基因编辑技术-BioVector NTCC质粒载体菌种细胞基因保藏中心
- 价 格:¥19820
- 货 号:pcDNA3.1-NgAgo, pET-NgAgo
- 产 地:北京
- BioVector NTCC典型培养物保藏中心
- 联系人:Dr.Xu, Biovector NTCC Inc.
电话:400-800-2947 工作微信:1843439339 (QQ同号)
邮件:Biovector@163.com
手机:18901268599
地址:北京
- 已注册
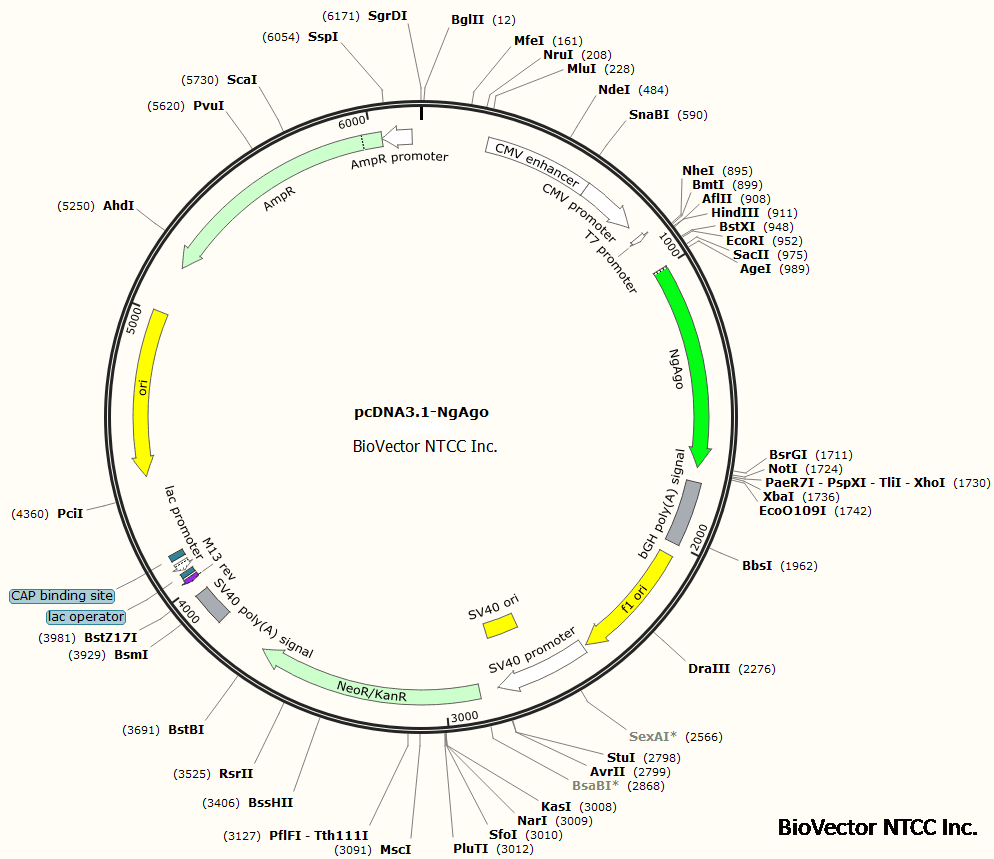
pcDNA3.1-NgAgo表达质粒载体
Map图谱:
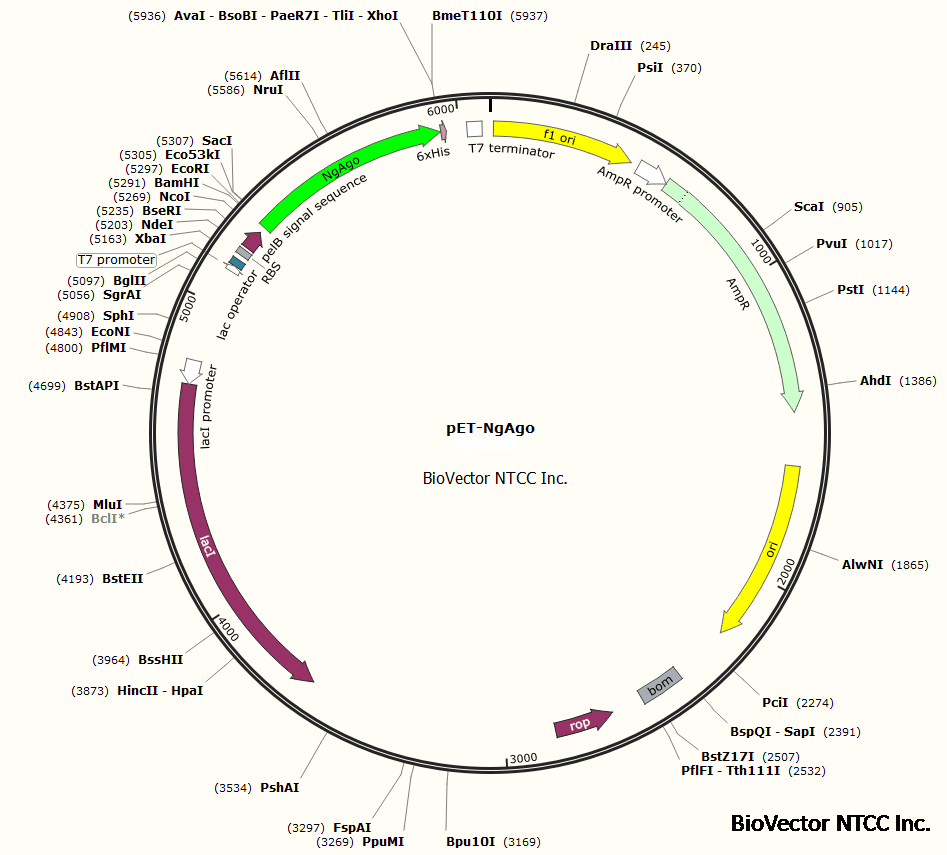
描述:
替代Cas9的新型基因编辑技术,Nature最新报道,表达Natronobacterium gregoryi Argonaute,规避了CAS9天然的弱点-脱靶效应,利用短链DNA作向导,真正实现了对基因组的任意位置进行切割,将基因编辑的可能性推入了更广泛的境地。
 该研究成果找到了对基因组位点编辑范围更广的基因编辑工具。该工具完全不同于以RNA为向导的CRISPR/Cas9基因编辑技术:这种从古细菌来源的Argonaute(简称NgAgo),利用短链DNA作向导,真正实现了对基因组的任意位置进行切割,将基因编辑的可能性推入了更广泛的境地。该项技术具有以下明确优势:
该研究成果找到了对基因组位点编辑范围更广的基因编辑工具。该工具完全不同于以RNA为向导的CRISPR/Cas9基因编辑技术:这种从古细菌来源的Argonaute(简称NgAgo),利用短链DNA作向导,真正实现了对基因组的任意位置进行切割,将基因编辑的可能性推入了更广泛的境地。该项技术具有以下明确优势:
1. 向导设计制作简便:可以像合成PCR引物一样合成短链单链DNA向导;向导可直接转染细胞和组织而无需构建向导表达载体。2. 可编辑基因组内任何位置:Cas9基因组的靶点选择受到PAM区和富含GC区的限制。而NgAgo对靶点选择没有限制,对基因组任何位置都能有效引入双链断裂。3,由于向导核酸是DNA而非RNA,因此避免了RNA易于形成复杂的二级结构而带来的失效或者脱靶效应。4,对游离于细胞核的DNA具有更高的切割效率。
与Cas9技术相比,Cas9需要19个配对的碱基,并且要求在需要编辑的基因组上这19个碱基后面必须紧邻一个符合一定特征的三碱基序列(PAM序列),这在一定程度上限制了gRNA的设计,而NgAgo–gDNA系统不需要PAM序列,拓宽了其设计范围。因此,从理论上讲,Cas9技术存在极限。
但是,NgAgo结合24个碱基的gDNA,这比Cas9的gRNA(19个碱基)要长5个碱基,理论上其精确性要提高1024(4的5次方)倍。DNA编辑相当于在一本书中的某个位置找到一个短单词将它替换成另一个长单词,这样就不容易出现误替换现象。举个例子,如果替换一篇文章中的the这样的简单单词,很容易把含有the三个字母的单词误替换了,如they,但是如果找一个更长的单词,如pneumonoultramicroscopy,就不容易出现误替换现象。
不仅如此,NgAgo–gDNA系统对向导序列-靶序列错配容忍度很低。gDNA上任何一个碱基的变换都会降低NgAgo的切割效率,如有三个错配则使其完全失活。这在另外一个机制上提高了NgAgo使用的精确性,特别是一些富含GC序列的地方,NgAgo系统比Cas9系统效率更高。NgAgo的gDNA需要5’端磷酸化的单链DNA (5p-ssDNA),这种形式的DNA在哺乳动物细胞中几乎不存在,这保证了NgAgo不会被内源的DNA序列错误带到不该去的地方。该系统的另外一个优点是5p-ssDNA是外源转化进细胞的,其时间和浓度可以非常精确地控制。而Cas9系统的gRNA是内源表达的质粒,难以精确地控制。
该技术可用于微生物、植物和动物的精准基因改造,以及乙肝、艾滋病或者一些遗传性疾病的“基因治疗”,在人类血液、器官的编辑和再造等方面具有重要意义,在医药,农业,畜牧等产业领域具有重要应用价值。
RNA 导向的核酸内切酶Cas9使得基因编辑成为了一种广泛应用的技术。然而,尽管科学家们已尝试多种方式对Cas9系统进行优化以提高它的效率和特异性,但该系统的实用性依然受到了一些因素的限制,包括其对导向-靶向(guide–target)错配的耐受性以及导向RNA相对容易形成二级结构等。
类似于Cas9,来自Argonaute蛋白家族的内切酶也可以利用寡核苷酸作为引导,降解侵入性的基因组。Argonautes在基因表达抑制以及抵御外来核酸方面起着关键的作用。
4月12日,发表在PNAS上的一项研究中,加州大学伯克利分校的研究人员揭示了一种Argonaute非典型的导向RNA特异性,论文的通讯作者是CRISPR先驱之一的Jennifer A. Doudna。
5月2日,最新发表在Nature Biotechnology上的一项研究中,河北科技大学以及浙江大学医学院的研究人员报告称,Natronobacterium gregoryi Argonaute(NgAgo)是一种DNA导向的可用于人类细胞基因编辑的核酸内切酶。NgAgo可结合约24个核苷酸大小的5′磷酸化单链导向 DNA(guide DNA ,gDNA),有效形成位点特异的DNA双链缺口。
据悉,CRISPR/Cas9的靶向特异性 是由两部分决定的,一部分是RNA嵌合体和靶DNA之间的碱基配对,另一部分是Cas9蛋白和一个短DNA基序(DNA motif)的结合,这个短DNA基序通常在靶DNA的3'末端发现,被称为前间区序列邻近基序(protospacer adjacent motif,PAM)[文献]。
与Cas9不同,NgAgo–gDNA系统不需要PAM,初步鉴定表明,该系统对导向-靶向(guide–target)错配耐受低,且对编辑富含G+C的基因组更加有效。据介绍,Cas9只存在于原核生物中,而Argonautes几乎存在于所有的有机体中。
此外,要想与Cas9正确绑定,导向RNA必须有3′RNA-RNA杂化结构,而与Argonaute绑定不需要导向分子有任何特定的二级结构。另一方面,Cas9只能切割PAM上游的序列,而Argonaute不需要靶标有特定的序列。作者们表示,Natronobacterium gregoryi Argonaute有望成为编辑哺乳动物基因组的精准有效的工具。
该技术的优势:
1. DNA 比RNA更稳定,更易操作。操做过RNA的都知道。
2. 削减了gRNA并非用于matching的冗余部分,精简了Primer,取消了PAM的限制,以及5' hairpin stemloop,而且直接用单链DNA。这些特质都完胜Crispr/Cas9
BioVector NTCC质粒载体菌种细胞基因保藏中心提供
- 公告/新闻